- I tipi di coronavirus sono tanti e diversi fra loro?
- Il Coronavirus muta facilmente?
- Ci sono particolari regioni più o meno soggette a mutazione?
Queste sono le domande che si sono posti due ricercatori italiani, Federico Giorgi e Carmine Ceraolo, in uno studio che è stato di recente pubblicato dalla rivista Journal of Medical Virology. Vediamo assieme quali sono state le loro scoperte.
I ceppi di coronavirus messi a confronto
I due ricercatori italiani hanno preso come base per il loro studio tutti i 56 diversi Coronavirus che sono stati finora isolati e sequenziati dall’inizio dell’epidemia nel mondo, estratti dai pazienti infettati dal Coronavirus: 43 identificati e provenienti dalla Cina, 2 dalla Thailandia, 5 dagli USA, 2 dall’Italia, 2 dalla Francia, 1 dalla Germania e 1 dall’Australia.
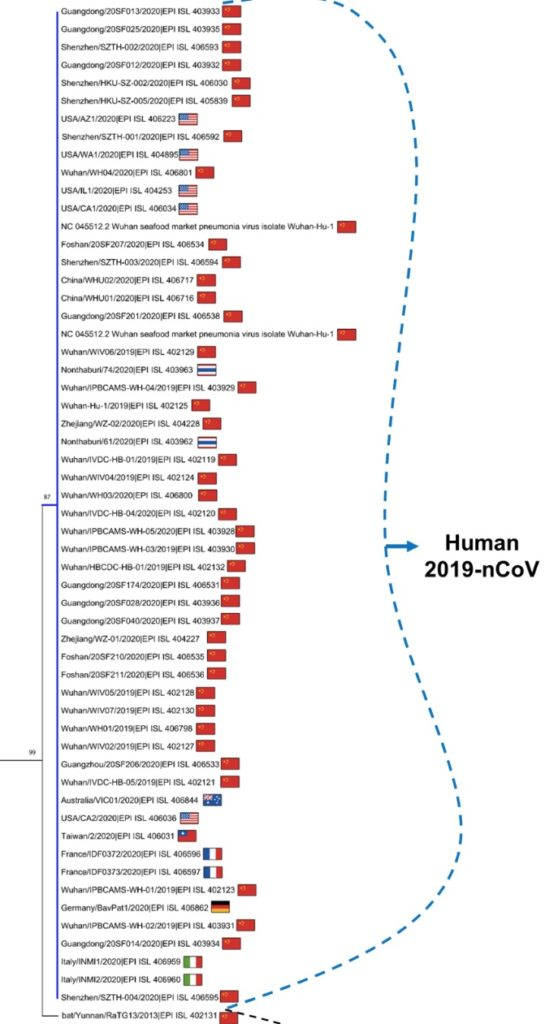
I risultati di questa analisi di un grande numero di dati ci raccontano che tutti questi genomi sono molto simili tra loro, anche provenienti da regioni molto differenti, tanto che le loro sequenze di codice sono sovrapponibili per più del 99%.
La somiglianza col virus del pipistrello
Questo ha permesso di poter condurre un’analisi del genoma del Coronavirus con quello degli altri Coronavirus animali, per capire a quale assomiglia maggiormente e quindi verosimilmente con quale virus animale risulti imparentato. Ebbene, il genoma del Coronavirus umano condivide le stesse sequenze del Coronavirus del pipistrello per il 96%. Nei confronti del virus della SARS, invece, ha una somiglianza soltanto dell’80%. Risulta prendere perciò maggior conferma quanto già ipotizzato riguardo l’origine del virus, ovvero che si sia sviluppato come una variante del virus del pipistrello in grado di colpire la specie umana.
Il fatto che il virus sia poco eterogeneo e poco mutabile autorizza ad essere piuttosto fiduciosi riguardo il fatto che le potenziali terapie che verranno utilizzate saranno efficaci nei confronti di tutti o quasi i ceppi virali.
Un punto di elevata variabilità
lo studio bolognese ha identificato inoltre un singolo punto di elevata variabilità delle proteine del virus. In particolare c’è una proteina di tipo accessorio del virus nella quale il cambiamento di un singolo aminoacido (da leucina a serina) può modificare la struttura della proteina stessa. Attualmente sono stati identificati due sottotipi di questa proteina ma non è ancora disponibile un modello che cerchi di comprendere a quali modificazioni strutturali della proteina corrispondano queste modifiche della sequenza genomica.
Prospettive
Questo studio è di grande interesse perchè ci ha fatto comprendere come attualmente i ceppi virali, per lo meno tutti quelli finora identificati, siano sostanzialmente simili tra loro e non vi siano enormi variabilità che possano farci temere una mutagenicità elevata. Questo è importante perchè eventuali future terapie e vaccini, stante la sostanziale uniformità del virus, dovrebbero essere efficaci su larga scala anzichè, come a volte può accadere, solo su alcuni ceppi virali.

Dott. Marco De Nardin