Identificate due diverse varianti di SARS-CoV-2
I coronavirus in natura vengono ospitati dai pipistrelli, dentro i quali sono liberi di modellare la loro evoluzione. Addirittura, è stato ipotizzato che la maggior parte dei coronavirus nell’uomo siano derivati dal serbatoio di pipistrelli presente in natura.
Proprio alla luce di questo, non sorprende che diversi team abbiano recentemente confermato la somiglianza genetica tra SARS-CoV-2 e un beta-coronavirus di pipistrello con cui condivide addirittura il 96,2% di genoma. Nello specifico, questa somiglianza è stata osservata tra SARS-CoV-2 ed un coronavirus di pipistrello correlato alla SARS, raccolto nella provincia dello Yunnan, in Cina.
Nonostante la sua correlazione con la SARS, si dimostra però più lontano evolutivamente rispetto ad essa, e molto più simile all’attuale coronavirus.
La via specifica di trasmissione dai serbatoi naturali agli umani rimane ad oggi poco chiara ed è ancora oggetto di ricerca, ma pochi giorni fa è stato pubblicata una pubblicazione (1) che rivela i risultati di uno studio comparato tra diversi virus isolati da pazienti positivi al COVID-19.
Per un approfondimento dal punto di vista virologico sulla struttura dei Coronavirus rimandiamo all’intervento integrale del Prof. Giorgio Palù, tratto dal primo evento-convegno online organizzato da Med4Care.
Lo studio
Per via della velocità con cui i virus possono mutare, infatti, è molto importante incentivare la ricerca sul monitoraggio dell’attuale SARS-CoV-2, ormai diffuso in oltre 20 paesi.
Lo studio (1) ha preso in considerazione 103 genomi di SARS-CoV-2 pubblicati in forma gratuita, dei quali hanno poi allineato le sequenze e identificato le varianti (o differenze) genetiche. Per facilitare la comprensione e l’analisi dei dati, ogni singolo virus è stato contrassegnato in base al luogo e alla data in cui è stato isolato.

In totale, sono state identificate mutazioni in 149 siti genici. Questo numero potrebbe spaventare, ma va detto che molte mutazioni di per sé non sono sufficienti per modificare la funzionalità del virus, mentre altre possono portare a cambiamenti di piccola entità. In buona sostanza, i diversi ceppi identificati sono molti, molti meno.
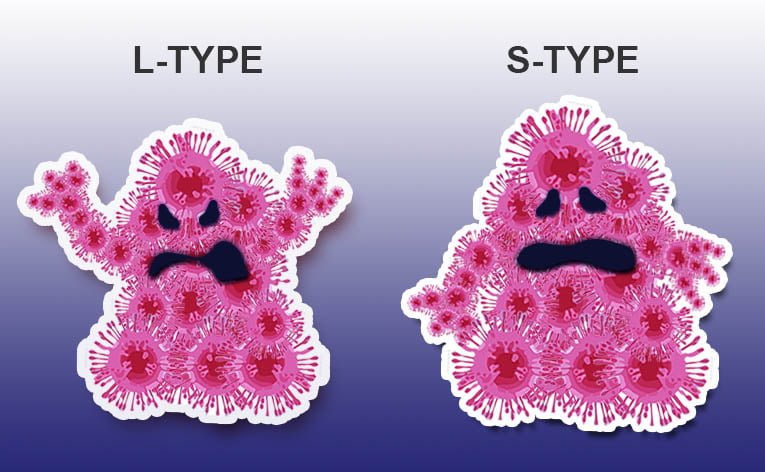
Le due varianti principali di SARS-CoV-2
Le analisi genetiche di popolazione dei 103 genomi di SARS-CoV-2 hanno indicato che questi virus si sono evoluti in due tipi principali, a cui sono stati dati i nomi di L ed S.
Il tipo L dall’inglese “large”, ossia grande, è il ceppo più diffuso: è stato rilevato nel 70% dei casi analizzati; il ceppo S, da “small” e quindi piccolo, è stato trovato nel 30% dei campioni ed è quello meno diffuso.
Sebbene il tipo L sia più prevalente del tipo S, è stato scoperto che il tipo S è a tutti gli effetti la versione ancestrale di SARS-CoV-2.
La distribuzione dei virus
La distribuzione dei due tipi di virus ha fatto capire ai ricercatori che L abbia una velocità di trasmissione più alta rispetto al tipo S. Inoltre, l’analisi della quantità di mutazioni nei diversi campioni ha indicato che il tipo L ha accumulato un numero significativamente maggiore di mutazioni rispetto al tipo S.
Sebbene il tipo L si sia evoluto recentemente, originato da mutazioni di mutazioni del più antico tipo S, esso si trasmette o si replica più velocemente nelle popolazioni umane. Questo spiegherebbe l’accumulo di un maggior numero di mutazioni rispetto al tipo S.
La conseguenza di questi risultati è che L potrebbe essere più aggressivo del tipo S proprio a causa della velocità di trasmissione e/o di replica potenzialmente più elevate.
Per verificare se i due tipi di SARS-CoV-2 presentavano differenze nelle distribuzioni in termini di tempo e spazio, gli studiosi hanno stratificato i virus in base alle localizzazioni e alle date in cui erano stati isolati. La distribuzione è stata:
- 27 virus isolati a Wuhan, di cui 26 (96,3%) erano di tipo L, e solo 1 (3,7%) era di tipo S;
- 73 virus isolati al di fuori di Wuhan, di cui 45 (61,6%) erano di tipo L e 28 (38,4 %) erano di tipo S.
Questo confronto suggerisce che il tipo L è significativamente più prevalente a Wuhan che in altri luoghi. Dati alla mano, il tipo L era più diffuso nelle prime fasi dell’epidemia a Wuhan, ma la sua frequenza è poi diminuita dopo il 7 gennaio 2020.
Alcune parziali conclusioni
Ma allora perché, se il tipo L è più aggressivo del tipo S, la frequenza del tipo L è diminuita rispetto al tipo S in altri luoghi dopo il breakout iniziale a Wuhan?
Una possibile spiegazione è che, da gennaio 2020, i governi centrale e locale del Sol Levante abbiano adottato misure di prevenzione e controllo così stringenti ed efficaci da provocare una selezione sui tipi di virus in circolo.
L’intervento dell’uomo, infatti, potrebbe aver causato una forte pressione selettiva contro il tipo L, potenzialmente più aggressivo e capace di diffondersi più rapidamente. Il tipo S, d’altra parte, potrebbe aver subito una pressione selettiva più debole proprio a causa dell’intervento umano, portando ad un aumento della sua abbondanza relativa tra i virus SARS-CoV-2.
L’ipotesi è dunque che le due varianti di SARS-CoV-2 potrebbero aver sperimentato diverse pressioni selettive a causa delle diverse caratteristiche epidemiologiche.
È da sottolineare che le analisi di cui sopra sono basate su genomi di SARS-CoV-2 raccolti da diverse località e punti temporali. Sono necessari dati genomici più completi per ulteriori test.
Altra nota importante, dicono i ricercatori, è che non è chiaro se il tipo L si sia evoluto dal tipo S negli umani o ancora prima, negli ospiti intermedi. Si deve inoltre comprendere se il tipo L sia più virulento del tipo S.
Per questi motivi, è di primaria importanza la necessità di ulteriori studi nell’immediato che combinino dati genomici, dati epidemiologici e resoconto dei sintomi clinici dei pazienti COVID-19.
AGGIORNAMENTO: per le ultime varianti covid, seguire questo link:

Fonti:
- (1) Xiaolu Tang, Changcheng Wu, Xiang Li, Yuhe Song, Xinmin Yao, Xinkai Wu, Yuange Duan, Hong Zhang, Yirong Wang, Zhaohui Qian, Jie Cui, Jian Lu, On the origin and continuing evolution of SARS-CoV-2, National Science Review.